Catégorie : Ressources
Logiciels de biométrie
Sylvain ARD – sylvain.ard@gmail.com
J’ai grandi dans les années 80 dans un petit village de Charente, Tusson. Enfant, je me suis passionné pour la nature : les insectes, les coquillages, les escargots, etc. Plus tard, vers l’âge de 12 ans, je me suis intéressé aux jeux vidéo, ce qui m’a amené à programmer mes propres jeux en autodidacte sur calculatrice. Des années ont passées et c’est à 20 ans que je me suis remis à mes deux anciennes passions : la nature (par le biais de macrophotographies) et l’informatique (par l’intermédiaire de la programmation sur ordinateur que j’ai apprise en autodidacte). A 24 ans j’ai obtenu un DAEU (équivalent du bac) et me suis inscrit en faculté d’informatique à l’Université de Poitiers. J’ai obtenu une licence pro en développement web et mobile. En 2014 j’ai décidé de créer mon autoentreprise.
J’étais en contact à l’époque avec Florian Mancet, président de Tela-Insecta (site francophone d‘entomologie) dont j’étais le programmeur bénévole. Pour certains de ses projets (exemple : identification de diptères par scans de leurs ailes ou application d’identification de criquets par leurs cris) et d’autres projets de naturalistes, j’ai réalisé qu’il fallait que j’acquières des compétences en traitement d’images et « reconnaissance de formes ». J’ai appris qu’il était possible de ne faire que la moitié de la licence ES et du master RTMA formant à ces disciplines à l’Université de Poitiers. La moitié, car l’autre était consacrée aux télécoms, ce qui ne m’était pas utile pour mon projet.
Plus tard, j’ai connu David Mercier, botaniste, durant un stage à la Station Universitaire du Limousin sur les Rubus (les ronces, dont il y a environ 2000 espèces en France). Durant ce stage, nous nous sommes mis d’accord pour que je développe un logiciel de biométrie de Rubus par ordinateur. Après quoi, il a été décidé que je conçoive durant mon stage de master, un logiciel de mesures automatique de Rubus sur parts d’herbier d’e-Recolnat (herbiers scannés du MNHN). Ce stage a été financé par des dons de botanistes. Cependant au sortir du stage et au vue la difficulté du problème (sur une part d’herbier tout est mélangé) mon programme n’était pas abouti.
Ultérieurement, j’ai développé différents programmes, fonctionnels, bien qu’imparfaits, de mesures de plantes fraîches avec des protocoles de scan plus stricts. Ensuite, David Mercier m’a fait connaître un rhodologue Michel Simon, qui réalisait déjà de gros travaux sur ordinateur (scans, mesures, etc.). J’ai effectué, pour lui aussi, des logiciels de biométrie automatique de Rosa sur scan (cf. figure 2). Voir note 1. Par la suite, toujours sur les conseils de David Mercier, j’ai commencé un programme de biométrie d’Alchémilles avec Thomas Weber, spécialiste suisse du genre. Seulement celui-ci étant trop occupé, il a abandonné le projet. Je suis en train de reprendre la tâche avec Martti Rajamäki et Rico Kaufmann, autres spécialistes. Par ailleurs, durant tout ce temps, j’avais également commencé un travail sur les Taraxacum avec Philippe Schwab et Tim Rich, taraxacologues mais laissé de côté pour l’instant. J’envisage aussi de créer un logiciel du même type pour les Hieracium. Tous ces projets sont bénévoles. J’ai quand même obtenu quelques contrats rémunérés durant ces années.
Pendant ce temps, j’ai également repris mon ouvrage sur la biométrie de Rubus sur parts d’herbier avec de nouvelles techniques comme le deep-learning. Tous ces programmes fonctionnent en effet de la même façon : les objets sont détectés par deep-learning puis mesurés par traitement d’images et géométrie. Les techniques de deep-learning que j’utilise sont principalement la segmentation sémantique et la segmentation d’instances. Dans ces deux techniques, on commence par dessiner les contours des objets à détecter manuellement sur quelques centaines d’images puis l’ordinateur « apprend » sur ces images pour ensuite « prédire » les contours de ces objets sur de nouvelles images. La différence entre les deux est que dans la segmentation d’instance les objets d’un même type (on dit d’une même classe) sont séparés contrairement à la segmentation sémantique (cf. figure 1). J’ai par exemple utilisé la segmentation d’instance pour les folioles, et la segmentation sémantique pour les tiges, pétioles et pétiolules (car plus difficilement séparables, puisque ramifiés. Je les sépare a posteriori par des techniques élaborées de traitement d’images telles que la squelettisation (cf. note 2) et des algorithmes pour trouver les nœuds et les branches (ceci fonctionne pour l’instant). Il existe d’autre techniques de deep-learning tels que la détection de points d’intérêt (comme les apex (i.e. « pointes ») et bases de folioles ou de feuilles) et la classification (on attribue une étiquette à l’image entière (cf. l’application mobile d’identification de plantes « PlantNet »).
Enfin en ce qui concerne le projet de biométrie sur parts d’herbier, je voudrais monter une cagnotte de 5000 euros pour rémunérer : un volontaire pour dessiner les points des apex et bases des folioles de 500 images de parts d’herbier (2000 euros), un expert en deep-learning que je connais pour améliorer le programme de segmentation sémantique de détection des tiges, pétioles, etc. (1000 euros) et enfin 2000 euros pour moi (pour le travail déjà accompli et à venir).
Pour en savoir plus vous pouvez me contacter à sylvain.ard@gmail.com

Figure 1. Segmentation sémantique à gauche, segmentation d’instances à droite
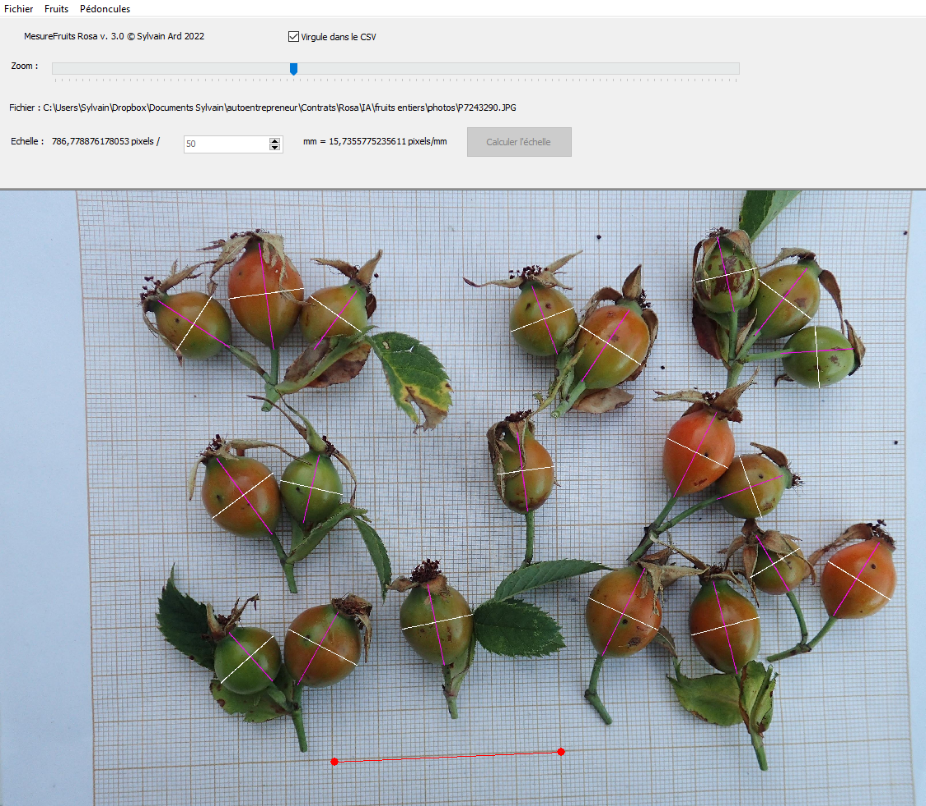
Figure 2. Programme de mesures de fruits de Rosa (églantier)
Note 1. On peut trouver tous ces logiciels de biométrie sur mon site Web : https://sylvain-ard.fr rubrique « Références » / « Projets d’IA ». Faites bien attention à installer auparavant toutes les dépendances nécessaires (Detectron2, Semantic2, Visual Studio etc., attention les dépendances ne sont pas les mêmes suivant les logiciels bien que certains en aient en commun) que vous trouverez sur la même page, et à bien regarder les recommandations en haut de la page (avertissement pour manque de certificat, bien lire la documentation avant d’installer, etc.).
Note 2. La squelettisation est une technique de traitement d’images consistant à amincir en « fils » d’un pixel de large, une forme.
Sites de données chorologiques
Lien vers la page des sites : docs.google.com/document/d/12bWz-aU0QwfM2pkMNa9-06vEjuHNr37WCwuWhRI0flk/edit?usp=sharing

